Auswirkungen von technologischem Fortschritt auf die Biosicherheit
Dieses Kapitel befasst sich mit Strategien zum Schutz vor biologischen Gefahren. Es untersucht zunächst eine Auswahl von Methoden zur Identifizierung von Krankheitserregern und gibt einen Überblick über ihre technischen Möglichkeiten, Mängel und zugrunde liegenden Prinzipien. Der zweite Teil taucht in eine breitere Analyse biologischer Angriffe ein und diskutiert dabei die mikrobielle Forensik und das Potenzial der gentechnischen Attribution.
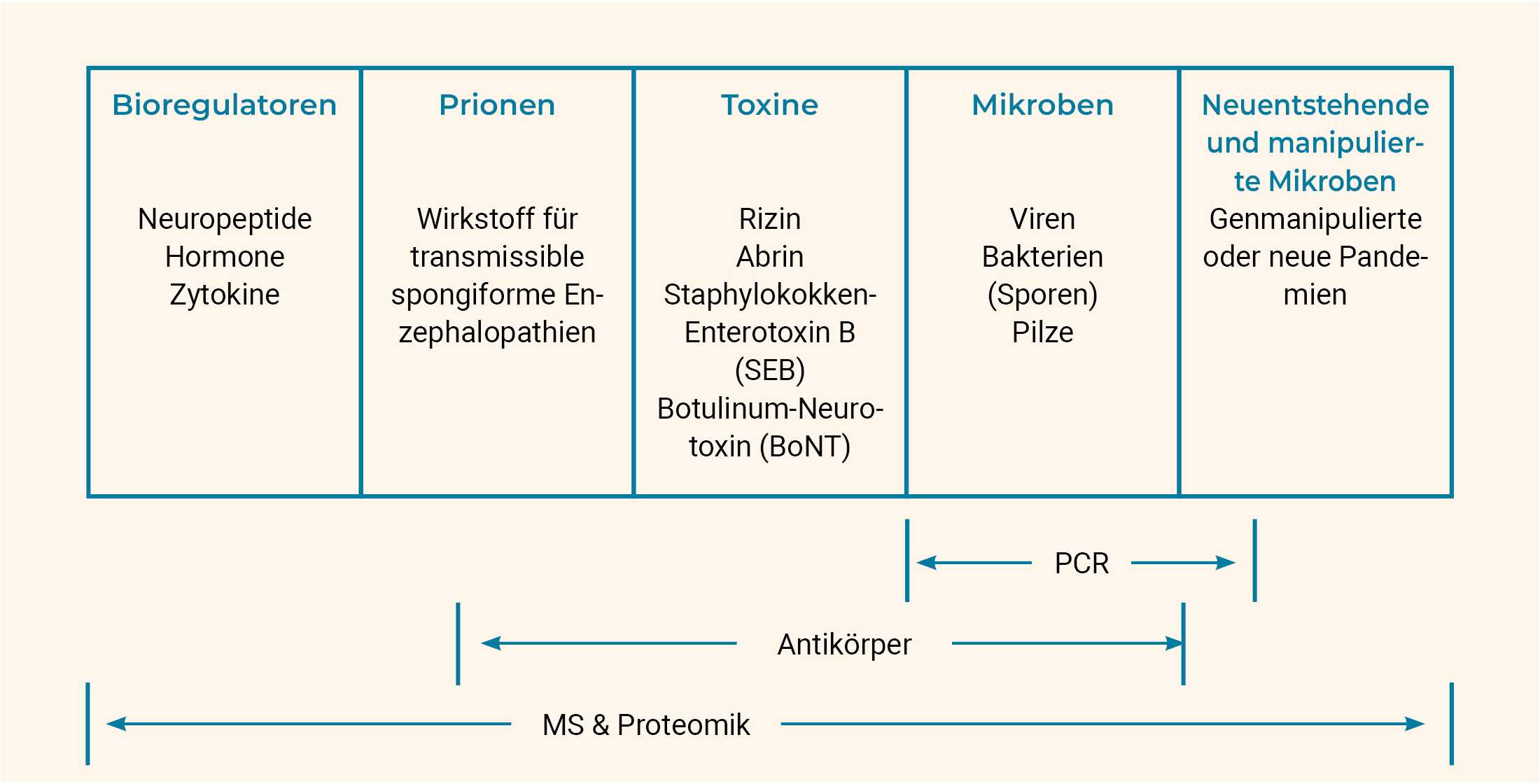
Ein zentrales Ziel der biologischen Abwehr (Biodefense) ist die Entwicklung schneller, empfindlicher und automatisierter Technologien, die in der Lage sind, biologische Kampfstoffe mit hoher Selektivität, Empfindlichkeit und Genauigkeit aufzuspüren und zu bestimmen. Der ideale Biomarker für den Nachweis von Krankheitserregern wäre einer, der für jeden biologischen Gefahrenstoff einzigartig und quantifizierbar ist. Die meisten Systeme zum Nachweis von Erregern verwenden Nukleinsäuren (z. B. DNA), Lipide und Proteine als primäre Elemente für die Erkennung. Gängige mikrobielle Stoffwechselprodukte wie flüchtige organische Verbindungen (VOCs) haben sich ebenfalls als vielversprechend für die Pathogenidentifizierung gezeigt.1
Die Detektionstechnologien konzentrieren sich auf eine Vielzahl von Methoden, die Oberflächenmerkmale, Marker (DNA-Sequenz mit bekannter physischer Position auf einem Chromosom), Proteomanalysen usw. umfassen. Während wie die Genomsequenzierung aufgrund ihrer hohen Sensitivität als guter Maßstab für den Nachweis von Krankheitserregern gelten, sind sie arbeitsintensiv, kostspielig und langsam, weshalb sich diese Methoden weniger an die Erfordernisse schneller Reaktionen anpassen lassen. Im Gegensatz dazu bieten molekulare Methoden wie die oder der Enzym-Immunoassay (ELISA) eine vergleichbare Sensitivität, Schnelligkeit und Genauigkeit. Allerdings erfordern molekulare Verfahren wie diese in der Regel hoch qualifiziertes Personal. Überdies ist zu beachten, dass Toxine mit diesen molekularen Strategien nicht identifiziert werden können, da sie keine DNA enthalten.
Molekulare Nachweismethoden
Pandemieviren und Schädlinge besitzen wie alle lebenden Organismen Genome, die aus Nukleinsäuren bestehen. Molekulare Nachweismethoden hängen von den unterschiedlichen Nukleinsäuremarkern ab, die für jeden biologischen Erreger spezifisch sind.
PCR (Polymerase-Kettenreaktion)
- Nach wie vor die bekannteste und meistgenutzte Technik
- Hauptvorteil ist ihre Spezifität, die sich aus der Einzigartigkeit des Genoms lebender Organismen ergibt.
- Höhere Sensitivität im Vergleich zu antikörperbasierten Methoden
- Ein erheblicher Nachteil ist die Unfähigkeit zur Unterscheidung lebender und toter Erreger.
- Um den Datendurchsatz zu erhöhen, müssten mehrere DNA-Zielsequenzen gleichzeitig nachgewiesen werden, indem mehrere Sätze von Primern in einer Reaktion kombiniert werden (sog. Multiplexing2).
Real-Time PCR (Echtzeit-PCR)
- Eine quantitative Methode, die den gleichzeitigen Nachweis und die Quantifizierung der Nukleinsäure in jeder Probe ermöglicht3
Wirksame Überwachungssysteme sollten unabhängig von spezifischen Phänotypen, Symptomprofilen oder Sequenzen funktionieren, die bei künftigen natürlichen Ausbrüchen oder Biowaffen erwartet werden. Angesichts der begrenzten Anzahl der bisher sequenzierten Viren kann es die Erkennungssysteme insensitiv für neue Pandemieerreger machen, wenn man sich auf bestehende Sequenzdatenbanken verlässt. Die oben genannten Methoden können eine Informationslücke bezüglich der genetischen Zusammensetzung eines Organismus offenlassen – ein kritischer Faktor für bioforensische Lösungen.
Sequenzbasierte Methoden
Next-Generation-Sequenzierung (NGS)
Mehrere Expert*innen sind der Ansicht, dass „Amerithrax“ (ein Wortgebilde aus „America“ und „Anthrax“, nach dem Namen des FBI-Falls4) eines der entscheidenden Ereignisse war, die zu einem erheblichen Anstieg der Forschungsmitteln für (Next-Generation-)Sequenzierungsanwendungen führten.5 Bei „Amerithrax“ handelte es sich um die biologischen Anschläge nach dem 11. September 2001, bei denen Briefe mit Milzbrandsporen an mehrere Medienbüros und Regierungsangestellte verschickt wurden. In den Untersuchungen wurde die zwar nicht direkt eingesetzt, aber die Forscher*innen verwendeten hochauflösende und vergleichende Genomik, um genetische Merkmale des Bacillus anthracis-Stammes aus den Briefen zu identifizieren. Durch die Möglichkeit, alle Nukleinsäuren in einer Probe für den Erregernachweis zu sequenzieren, sind keine Vorkenntnisse über den Erreger nötig, wie sie normalerweise für gezielte molekulare Tests (wie Echtzeit-PCR) erforderlich sind. Der Prozess beginnt mit der Extraktion der Genom-DNA aus der Probe. Diese DNA wird dann in eine umgewandelt, die anschließend sequenziert wird. Die sich daraus ergebenden Sequenzdaten werden mit bekannten Referenzsequenzen von Mikroorganismen verglichen, von denen angenommen wird, dass sie in der Probe vorkommen.

- Next-Generation-Sequenzierung (Next Generation Sequencing, NGS) umfasst mehrere Technologien, die zur Sequenzierung der Nukleotide in vollständigen Genomen oder in bestimmten DNA- oder RNA-Regionen eingesetzt werden.
- Die NGS-Plattformen bieten eine massiv parallele Sequenzierung, entweder von klonal amplifizierten oder einzelnen DNA-Molekülen.
- NGS sequenziert DNA durch wiederholte Zyklen von polymerasegesteuerten Nukleotidverlängerungen, wobei in einem einzigen Durchlauf Hunderte von Megabasen bis hin zu Gigabasen an Sequenzdaten erzeugt werden.
- Bei NGS-Experimenten werden riesige Datenmengen erzeugt, die eine Herausforderung für die Datenverarbeitung, -speicherung und vor allem für die Analyse darstellen.
- NGS hat die Erforschung der mikrobiellen Vielfalt sowohl in der Umwelt als auch in klinischen Umgebungen revolutioniert.
Metagenomische Sequenzierung
Bei der metagenomischen Sequenzierung werden alle mikrobiellen und Wirtsnukleinsäuren in einer Probe ohne vorherige Selektion (kulturunabhängig) sequenziert. Dies geschieht in der Regel mit der sogenannten .7 Das Endziel der Metagenomik besteht darin, die mikrobielle Überwachung zu revolutionieren und Initiativen im Bereich der öffentlichen Gesundheit durch proaktives Screening auf Bedrohungen zu stärken.
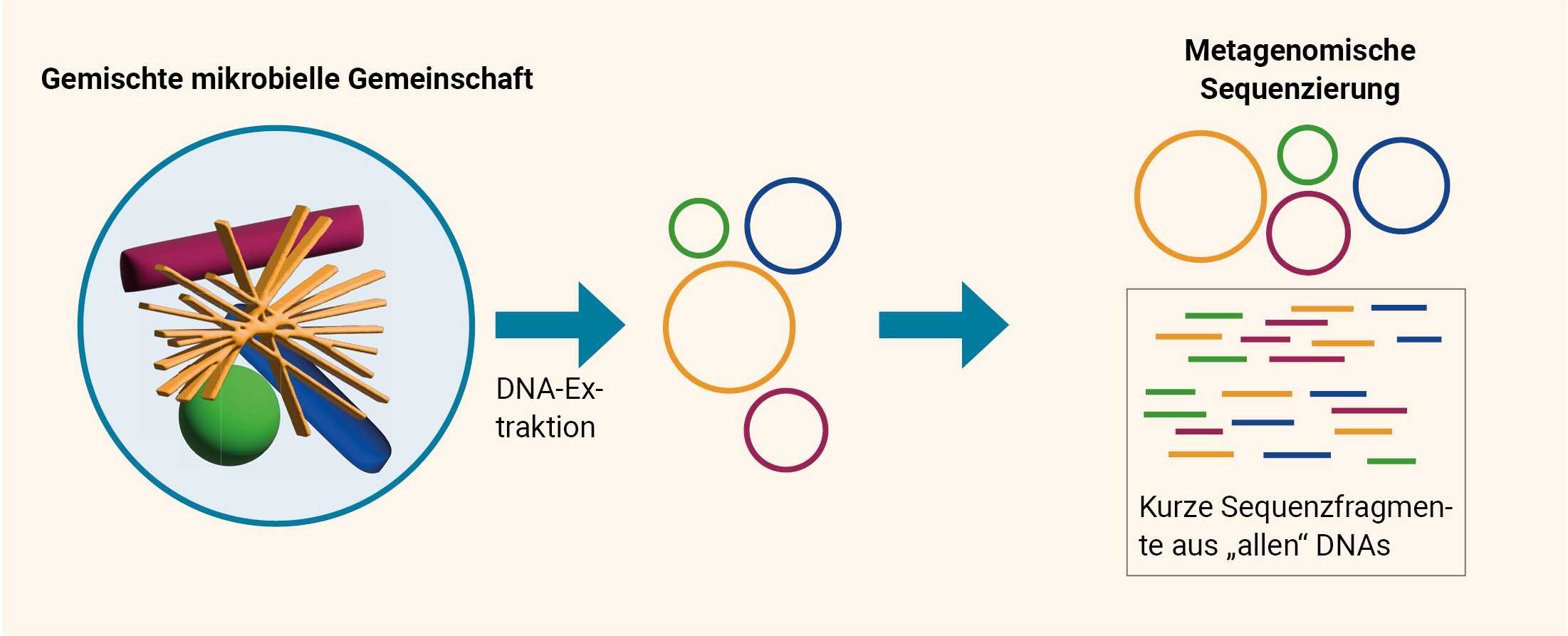
Nach mehreren Runden der Fragmentierung und Sequenzierung ergeben sich mehrere überlappende Reads für die Ziel-DNA. Die überlappenden Enden der verschiedenen Reads werden verwendet, um sie zu einer kontinuierlichen Sequenz zusammenzusetzen.
- Das metagenomische NGS ist unvoreingenommen und daher erregerunabhängig. Es werden keine spezifischen Primer oder Sonden verwendet, so dass jeder Erreger nachgewiesen werden kann.
- Die überwiegende Mehrheit der sequenzierten Reads stammt aus dem menschlichen Wirt, was das Verfahren rechnerisch komplexer macht. Die Identifizierung von Krankheitserregern in diesen metagenomischen Bibliotheken wird zu einer schwierigen Aufgabe.
Sequenzierung von Abwässern
DNA- und RNA-Fragmente von Krankheitserregern (und anderen lebenden Organismen) werden immer wieder in Gewässern auf der ganzen Welt entdeckt. Neue Techniken ermöglichen es, sie zu identifizieren.9 So wurde beispielsweise SARS-CoV-2 einige Tage vor der Meldung der ersten klinischen Fälle im Abwasser nahegelegener Gemeinden nachgewiesen.10 Anhand der Abwassersequenzierung konnten auch Varianten des Virus nachgewiesen und identifiziert werden.11
Dabei werden Proben vor der Abwasseraufbereitung entnommen und in Labors analysiert, um die Art und die Menge an Krankheitserregern in einer Gemeinde zu ermitteln. Next-Generation-Sequenzierung kann auch für diese Analyse eingesetzt werden und bietet Angestellten im öffentlichen Gesundheitswesen eine schnelle und umfassende Methode, um Infektionen großflächig zu überwachen. Die Überwachung von Krankheitserregern ist eine regelmäßige Praxis im öffentlichen Gesundheitswesen, die in verschiedenen Einrichtungen wie Krankenhäusern, Wasseraufbereitungsanlagen und landwirtschaftlichen Betrieben durchgeführt wird, wobei der Schwerpunkt auf Umgebungen liegt, die als anfällig für potenzielle Krankheitsausbrüche erachtet werden. Der Hauptvorteil der Abwassersequenzierung zum Nachweis von Humanpathogenen besteht darin, dass die Privatsphäre gewahrt wird.12 Zudem ermöglicht sie es, bekannte und neu auftretende Krankheitserreger zu beobachten sowie frühzeitig Risikominderungsstrategien für den Gesundheitsschutz von Menschen und Tieren anzuwenden.
Proteomik- und Lipidomik-Methoden
Die hat das Verständnis der Virulenz, der Struktur und der Pathogenese von Krankheitserregern verbessert, was bei der Diagnose von Krankheiten und der Entwicklung von Impfstoffen hilft. Die Proteomik wurde eingesetzt, um bakterielle und virale Proteine zu untersuchen, die für die Übertragung von Krankheiten bei Menschen und Tieren verantwortlich sind – Membranproteine, Oberflächenproteine und sekretierte Proteine sind der Schlüssel zur Pathogenität. Diese Proteine, die verschiedene kritische Funktionen erfüllen, z. B. als Enzyme, Transporter und Rezeptoren, sind für den Ausbruch und das Fortschreiten von Krankheiten von entscheidender Bedeutung.
Die Analyse proteomischer Variationen in der Pathogenese von Bakterien und Viren ermöglicht es, verschiedene Stämmen hinsichtlich der Schwere der Infektion, der Auswirkungen auf die Umwelt und der Folgen genetischer Veränderungen zu vergleichen.
Die ist der umfassendste Ansatz zur quantitativen Profilerkennung von Proteinen, ihren Wechselwirkungen und Veränderungen. Die Verfügbarkeit verschiedener Massenspektrometrie-Plattformen in Verbindung mit mikrobiellen Genomdatenbanken eröffnet neue Möglichkeiten.14 Die Fähigkeit der Massenspektrometrie, mehrere Analyten wie Luft, Wasser, Nährmedien, Körperflüssigkeiten und Lebensmittel gleichzeitig in komplexen Matrizen zu analysieren, verschafft einen wesentlichen Vorteil dabei, biologische Bedrohungen zu erkennen und zu bekämpfen. MS-Technologien werden in Verbindung mit anderen Methoden auch häufig in der chemischen Forensik zu Ermittlungszwecken eingesetzt.
- MS ist sowohl schnell als auch sensitiv – ein typisches Experiment, einschließlich Probenentnahme und -vorbereitung, kann innerhalb von Minuten durchgeführt werden, während herkömmliche mikrobiologische oder toxikologische Tests Tage in Anspruch nehmen.
- Was die Sensitivität betrifft, so kann MS biologische Gefahren in Proben nachweisen, die eine geringe Menge intakter Organismen oder eines Toxins enthalten.
Beispiele für MS-Methoden zur Identifizierung von Mikroorganismen
- Plattformen wie die Matrix-Assistierte Laser-Desorptions-Ionisations-Flugzeit-Massenspektrometrie (MALDI-TOF-MS) und die Flüssigchromatographie-Tandem-Massenspektrometrie (LC-MS/MS) werden zunehmend in der mikrobiologischen Forschung, insbesondere zu diagnostischen Zwecken, eingesetzt. Sie basieren auf Techniken des oder der Peptidsequenzierung.15 Die U.S. Food and Drug Administration hat auch MALDI-TOF-MS-basierte Plattformen wie den MALDI Biotyper (MBT) zugelassen.16 Aufgrund der schnellen Analysezeit und der geringen Kosten für die Probenverarbeitung ist der MBT eine zuverlässige Option für die Identifizierung von Mikroorganismen. Indem es den einzigartigen proteomischen Fingerabdruck eines Organismus bestimmt, vergleicht MALDI-TOF MS diesen mit einer umfassenden und ständig aktualisierten Referenzbibliothek, um die Identität des Organismus genau zu bestimmen.
- In einigen Fällen sollte eine mikrobielle Charakterisierung auf der Grundlage anderer Biomarker wie Lipide in Betracht gezogen werden.17 In diesem Zusammenhang eröffnen die Fortschritte bei einer anderen MS-Technologie – der Umgebungsionisations-Massenspektrometrie (AIMS) – neue Perspektiven für den schnellen und präzisen Echtzeitnachweis von Spuren neu aufkommender pathogener Mikroorganismen und Toxine. Diese Methode erfordert wenig oder keine Vorbereitung der Probe, was einen großen Vorteil darstellt.18 Die „Umgebungsionisierung“ ist eine Ionisierungsmethode, bei der Ionen in der Gasphase direkt in Freiluft erzeugt werden, wodurch eine chromatografische Trennung überflüssig wird und die vorherige Probenvorbereitung erheblich verringert wird.19 Dies ermöglicht eine nahezu sofortige Charakterisierung mikrobieller Spezies.
- Unternehmen, die sich auf die biologische Abwehr und den Nachweis von Krankheitserregern spezialisiert haben, wie BioFlyte,20 stellen tragbare Massenspektrometer für den Nachweis von Aerosolen her. Diese MS-Geräte können Substanzen in Aerosolen nachweisen, darunter Viren, Bakterien, Sporen, Opioide und andere Stoffe. Zu den neusten Entwicklungen gehören Systeme zum Nachweis von Atemwegserregern in Innenräumen.
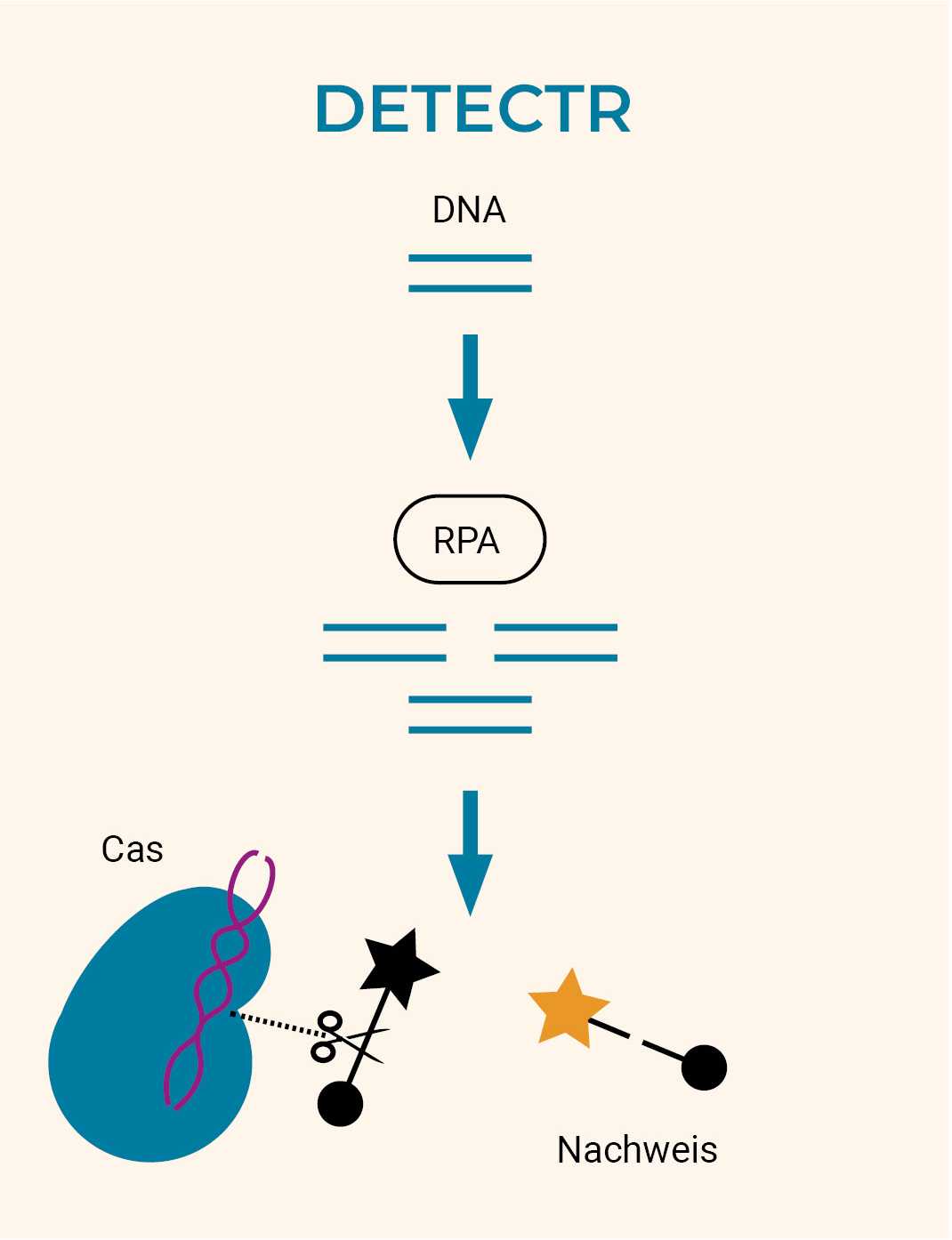
CRISPR-Cas-Systeme für die Diagnostik und den Nachweis von Krankheitserregern
Neben hochspezifischen Veränderungen menschlicher Gene können auch einen schnelleren und genaueren Nachweis von viralen und bakteriellen Nukleinsäuren bieten. Zwei analoge Diagnosesysteme, bekannt als DETECTR und SHERLOCK, wurden entwickelt, um RNA- bzw. DNA-Sequenzen nachzuweisen.21 Virale oder bakterielle RNA oder DNA wird amplifiziert und dann mit einem CRISPR/Cas-System gemischt, das die gewünschte Sequenz identifiziert und schneidet. Nach der Spaltung der Zielsequenzen beginnt das Cas-Protein, alle anderen DNAs oder RNAs in der Lösung zu spalten. Wenn also eine Reportersequenz eingeführt wird, deren Fluoreszenzsignal durch ein anderes Molekül unterdrückt wird, spaltet Cas auch diesen Komplex und erzeugt ein Fluoreszenzsignal, das gemessen werden kann.
Beispielsweise entwickelt Mammoth Biosciences22 Diagnostika, Therapeutika und Proteinforschung auf der Grundlage der CRISPR-Technologie, die hohe Genauigkeit, Vielseitigkeit (Pathogen-Agnostizismus) und einfache Anwendung verspricht.23 CRISPR-Cas-Proteine gelten aufgrund ihrer Schnelligkeit und Genauigkeit als ideale Werkzeuge für den Nachweis von Nukleinsäuren, da sie eine Leit-RNA (gRNA) verwenden, um DNA- oder RNA-Sequenzen auf natürliche Weise zu adressieren. Gewünschte Genome können durch einfache Veränderung der gRNA-Zielregion anvisiert werden, wie die schnelle Anpassung der CRISPR-basierten Diagnostik während der COVID-19-Pandemie gezeigt hat.24
Mikrobielle Forensik und Attribution von Gentechnik
Motivation für mikrobielle Forensik
Obwohl Mikroben schon seit Jahrhunderten als Waffen eingesetzt werden, lösten die Milzbrandanschläge in den USA im Jahr 2001 in der Öffentlichkeit große Angst aus.26 Diese Ereignisse machten deutlich, dass eine Infrastruktur von Analyseinstrumenten und Wissensgrundlagen benötigt wird, die zügig Hinweise für die Ermittlungen liefert, die Identifizierung von Verantwortlichen (Attribution) unterstützt, Aufschluss über die Quelle des Erregers gibt und Details darüber aufdeckt, wie und wo die Waffe hergestellt wurde. Die mikrobielle Forensik hatte sich zwar bereits vor den 2000er Jahren als wissenschaftliches Gebiet entwickelt, erfuhr jedoch durch die Amerithrax-Ereignisse einen enormen Aufschwung. Dieser Zweig der forensischen Wissenschaft befasst sich mit der Untersuchung von Beweismaterial, das aus bioterroristischen Anschlägen, biologischen Verbrechen oder der unbeabsichtigten Freisetzung von Mikroorganismen oder Giftstoffen stammt, um deren Ursprung zu ermitteln. Der Schwerpunkt dabei liegt darauf, die Herkunft von Krankheitserregern zurückzuverfolgen, um die Strafverfolgung der für ihre Verbreitung verantwortlichen Personen zu unterstützen. Ziel ist es, eine Probe unklarer Herkunft mit größtmöglicher wissenschaftlicher Gewissheit einer oder mehreren bekannten Quellen genau zuzuordnen und gleichzeitig andere Quellen auszuschließen. Die globale COVID-19-Pandemie und die Ungewissheit über die Herkunft des Virus unterstreichen sowohl die Dringlichkeit als auch die potenziellen geopolitischen Auswirkungen dieser Technologien. Biologische Ereignisse zu untersuchen und einer Quelle zuzuordnen stellt eine Herausforderung dar, und selbst vier Jahre nach der COVID-19-Pandemie sind sich die US-Geheimdienste über den Ursprung des Virus nicht einig.
Gentechnische Attribution
Das Risiko, dass die Biologie missbraucht wird und weitreichende Schäden verursacht, steigt mit dem rasanten technologischen Fortschritt. Dies wiederum erschwert die Herausforderung der Attribution – der Feststellung, ob der verursachende Organismus gentechnisch verändert wurde, und falls ja, der Identifizierung der Verantwortlichen. Die Attribution kann drei wesentliche Vorteile für die Sicherheit bringen. Erstens hilft die Kenntnis der verantwortlichen Personen, ihrer Motive und Fähigkeiten, die Reaktionsmaßnahmen zu justieren. Zweitens ermöglicht Attribution, die verantwortlichen Parteien zu identifizieren, so dass angemessene zivil-, strafrechtliche oder diplomatische Sanktionen angewendet werden können. Drittens wirkt eine wirksame Attribution in Verbindung mit entschlossenen Maßnahmen, um die Verantwortlichen zur Rechenschaft zu ziehen, als Abschreckung gegen rücksichtsloses oder schädliches Verhalten.27 Das Risiko erhöht sich für diejenigen, die an illegalen Biowaffen-, Bioterrorismus- oder Bioproliferationsaktivitäten beteiligt sind, wenn sie gefasst und zur Rechenschaft gezogen werden oder wenn die realistische Aussicht darauf besteht.
In der mikrobiellen Forensik wird, wann immer dies möglich ist, eine detaillierte Identifizierung des mikrobiellen Erregers bis auf Substammebene (ein Stamm, der von dem charakterisierten Stamm abstammt, sich aber von ihm unterscheidet) angestrebt. Im Labor vorgenommene Veränderungen an Mikroorganismen, wie Klonierung und gentechnische Veränderung, hinterlassen nachweisbare Spuren in ihren Genomen.28 Diese genetischen Marker dienen zusammen mit beobachtbaren Merkmalen als Beweis dafür, dass gefährliche Erreger absichtlich erzeugt und verbreitet wurden, um Schäden anzurichten. Ein Merkmal in mikrobieller DNA, das für die Analyse der molekularen Phylogenie nützlich ist, sind . SNPs sind als genetische Marker sehr stabil und informativ für Stammbaumstudien, obwohl sie für die Identifizierung einzelner Isolate nur eine geringe Aussagekraft haben.
In einem Genom können sich verräterische Spuren gentechnischer Veränderung auf verschiedene Weise manifestieren. Der Entscheidungsprozess des Genforschers wird von verschiedenen Faktoren beeinflusst, wie z. B. der Ausbildung, früheren Erfahrungen, etablierten Praktiken und der Verfügbarkeit von Ressourcen. Diese gesammelten Entscheidungen bilden eine unverwechselbare „methodische Signatur“, die es ermöglicht, die Design-Entscheidungen auf den wahrscheinlichen Designer zurückzuführen. Dies kann das Vorhandensein fremden genetischen Materials in einer bestimmten Sequenz ebenso einschließen wie das unerwartete Auftauchen etwa von Verdopplungen, Einfügungen (Insertionen) oder Streichungen (Deletionen) von Basen. Darüber hinaus können Sequenzen, die für Antibiotikaresistenzen kodieren, und kurze Abschnitte, die als „Narben“ bekannt sind und auf Veränderungen der DNA-Sequenz hinweisen, als Hinweise für die Entdeckung von Manipulationen dienen. Obwohl es keine endgültige Liste aller „Engineering-Sequenzen“ gibt, bieten Plattformen wie Addgene29 umfangreiche Datenbanken mit molekularen Werkzeugen, die für DNA-Manipulationen im Labor genutzt werden. Der Addgene-Datensatz, ein umfassendes und sorgfältig kuratiertes Repositorium von Plasmidsequenzen zusammen mit Informationen zu den Autor*innen, dient als vereinfachte Darstellung der Gentechnik-Attribution (Genetic Engineering Attribution, GEA). Während dieser Datensatz für die Weiterentwicklung der GEA-Methoden unverzichtbar war, müssen praktische Attributionsmodelle letztlich über diesen ursprünglichen Rahmen hinausgehen und ein breiteres Spektrum von Szenarien abdecken.
Einige Expert*innen haben die Befürchtung geäußert, dass das Wissen über die gentechnische Attribution die politische Komplexität erhöhen könnte, da ein böswilliger Akteur absichtlich gentechnische Merkmale auswählen könnte, um ein anderes Labor oder ein anderes Land fälschlich zu belasten. Darüber hinaus könnte der Grad an Vertrauen, der erforderlich ist, um ein bestimmtes Labor als Täter zu benennen, komplexe diplomatische Entscheidungen beinhalten.
Auch wenn die GEA-Methoden noch in den Kinderschuhen stecken und die Attribution eine komplexe Aufgabe bleibt, könnten sie dennoch eine wissenschaftliche Grundlage für die BWÜ-Diskussionen zu Verifikation und Compliance bieten.
- Filipiak, W., Ager, C., & Troppmair, J. (2017). Predicting the future from the past: Volatile markers for respiratory infections. European Respiratory Journal, 49(5), 1700264. https://doi.org/10.1183/13993003.00264-2017 ↩
- Kreitmann, L., Miglietta, L., Xu, K., Malpartida-Cardenas, K., D’Souza, G., Kaforou, M., Brengel-Pesce, K., Drazek, L., Holmes, A., & Rodriguez-Manzano, J. (2023). Next-generation molecular diagnostics: Leveraging digital technologies to enhance multiplexing in real-time PCR. TrAC Trends in Analytical Chemistry, 160, 116963. https://doi.org/10.1016/j.trac.2023.116963 ↩
- Dung, T. T. N., Phat, V. V., Vinh, C., Lan, N. P. H., Phuong, N. L. N., Ngan, L. T. Q., Thwaites, G., Thwaites, L., Rabaa, M., Nguyen, A. T. K., & Duy, P. T. (2024, 7. Februar). Development and validation of multiplex real-time PCR for simultaneous detection of six bacterial pathogens causing lower respiratory tract infections and antimicrobial resistance genes. BMC Infectious Diseases, 24(1), 164. https://doi.org/10.1186/s12879-024-09028-2 ↩
- The United States Department of Justice. (2010, 19. Februar). Amerithrax Investigative Summary. The United States Department of Justice. https://www.justice.gov/archive/amerithrax/docs/amx-investigative-summary.pdf ↩
- Rasko, D. A., Worsham, P. L., Abshire, T. G., Stanley, S. T., Bannan, J. D., Wilson, M. R., Langham, R. J., Decker, R. S., Jiang, L., Read, T. D., Phillippy, A. M., Salzberg, S. L., Pop, M., Van Ert, M. N., Kenefic, L. J., Keim, P. S., Fraser-Liggett, C. M., & Ravel, J. (2011, 7. März). Bacillus anthracis comparative genome analysis in support of the Amerithrax investigation. Proceedings of the National Academy of Sciences, 108(12), 5027–5032. https://doi.org/10.1073/pnas.1016657108 ↩
- Minogue, T. D., Koehler, J. W., Stefan, C. P., & Conrad, T. A. (2019, 1. März). Next-Generation Sequencing for Biodefense: Biothreat Detection, Forensics, and the Clinic. Clinical Chemistry, 65(3), 383–392. https://doi.org/10.1373/clinchem.2016.266536 ↩
- Quince, C., Walker, A. W., Simpson, J. T., Loman, N. J., & Segata, N. (2017, 12. September). Shotgun metagenomics, from sampling to analysis. Nature Biotechnology, 35(9), 833–844. https://doi.org/10.1038/nbt.3935 ↩
- Lee, M. (2019, 29. September). Happy Belly Bioinformatics: An open-source resource dedicated to helping biologists utilize bioinformatics. Journal of Open Source Education, 2(19), 53. https://doi.org/10.21105/jose.000539 ↩
- The Nucleic Acid Observatory Consortium. (2021). A Global Nucleic Acid Observatory for Biodefense and Planetary Health (arXiv:2108.02678). arXiv. https://doi.org/10.48550/ARXIV.2108.02678 ↩
- Peccia, J., Zulli, A., Brackney, D. E., Grubaugh, N. D., Kaplan, E. H., Casanovas-Massana, A., Ko, A. I., Malik, A. A., Wang, D., Wang, M., Warren, J. L., Weinberger, D. M., Arnold, W., & Omer, S. B. (2020, 18. September). Measurement of SARS-CoV-2 RNA in wastewater tracks community infection dynamics. Nature Biotechnology, 38(10), 1164–1167. https://doi.org/10.1038/s41587-020-0684-z ↩
- Crits-Christoph, A., Kantor, R. S., Olm, M. R., Whitney, O. N., Al-Shayeb, B., Lou, Y. C., Flamholz, A., Kennedy, L. C., Greenwald, H., Hinkle, A., Hetzel, J., Spitzer, S., Koble, J., Tan, A., Hyde, F., Schroth, G., Kuersten, S., Banfield, J. F., & Nelson, K. L. (2021, 19. Januar). Genome Sequencing of Sewage Detects Regionally Prevalent SARS-CoV-2 Variants. mBio, 12(1), e02703-20. https://doi.org/10.1128/mBio.02703-20; Pérez-Cataluña, A., Chiner-Oms, Á., Cuevas-Ferrando, E., Díaz-Reolid, A., Falcó, I., Randazzo, W., Girón-Guzmán, I., Allende, A., Bracho, M. A., Comas, I., & Sánchez, G. (2021, 10. Februar). Detection Of Genomic Variants Of SARS-CoV-2 Circulating In Wastewater By High-Throughput Sequencing. medRxiv. https://doi.org/10.1101/2021.02.08.21251355 ↩
- The Nucleic Acid Observatory Consortium, 2021. ↩
- Demirev, P. A., & Fenselau, C. (2008). Mass spectrometry in biodefense. Journal of Mass Spectrometry, 43(11), 1441–1457. https://doi.org/10.1002/jms.1474 ↩
- Drake, R. R., Deng, Y., Schwegler, E. E., & Gravenstein, S. (2005). Proteomics for biodefense applications: Progress and opportunities. Expert Review of Proteomics, 2(2), 203–213. https://doi.org/10.1586/14789450.2.2.203 ↩
- Rajoria, S., Sabna, S., Babele, P., Kumar, R. B., Kamboj, D. V., Kumar, S., & Alam, S. I. (2020, 10. Februar). Elucidation of protein biomarkers for verification of selected biological warfare agents using tandem mass spectrometry. Scientific Reports, 10(1), 2205. https://doi.org/10.1038/s41598-020-59156-3 ↩
- Bruker. (2024). MALDI Biotyper (MBT). https://www.bruker.com/en/products-and-solutions/microbiology-and-diagnostics/microbial-identification.html ↩
- Su, H., Jiang, Z.-H., Chiou, S.-F., Shiea, J., Wu, D.-C., Tseng, S.-P., Jain, S.-H., Chang, C.-Y., & Lu, P.-L. (2022, 26. April). Rapid Characterization of Bacterial Lipids with Ambient Ionization Mass Spectrometry for Species Differentiation. Molecules, 27(9), 2772. https://doi.org/10.3390/molecules27092772 ↩
- Ferreira, C. R., Yannell, K. E., Jarmusch, A. K., Pirro, V., Ouyang, Z., & Cooks, R. G. (2016, 1. Januar). Ambient Ionization Mass Spectrometry for Point-of-Care Diagnostics and Other Clinical Measurements. Clinical Chemistry, 62(1), 99–110. https://doi.org/10.1373/clinchem.2014.237164 ↩
- Venter, A., Nefliu, M., & Graham Cooks, R. (2008). Ambient desorption ionization mass spectrometry. TrAC Trends in Analytical Chemistry, 27(4), 284–290. https://doi.org/10.1016/j.trac.2008.01.010 ↩
- Bioflyte. (2024). BioFlyte — Biosecurity at Scale. https://www.bioflyte.com/ ↩
- Für eine Zusammenfassung siehe: Kocak, D. D. & Gersbach, C. A. (2018). From CRISPR scissors to virus sensors. Nature, 557, 168–169. https://doi.org/10.1038/d41586-018-04975-8 ↩
- Mammoth Biosciences. (2024). Mammoth Biosciences. https://mammoth.bio/ ↩
- Verosloff, M. S., Shapiro, S. J., Hawkins, E. M., Alpay, E., Verma, D., Stanfield, E. G., Kreindler, L., Jain, S., McKay, B., Hubbell, S. A., Hendriks, C. G., Blizard, B. A., Broughton, J. P., & Chen, J. S. (2022). CRISPR‐Cas enzymes: The toolkit revolutionizing diagnostics. Biotechnology Journal, 17(7), 2100304. https://doi.org/10.1002/biot.202100304 ↩
- Broughton, J. P., Deng, X., Yu, G., Fasching, C. L., Servellita, V., Singh, J., Miao, X., Streithorst, J. A., Granados, A., Sotomayor-Gonzalez, A., Zorn, K., Gopez, A., Hsu, E., Gu, W., Miller, S., Pan, C.-Y., Guevara, H., Wadford, D. A., Chen, J. S., & Chiu, C. Y. (2020, 16. April). CRISPR–Cas12-based detection of SARS-CoV-2. Nature Biotechnology, 38(7), 870–874. https://doi.org/10.1038/s41587-020-0513-4 ↩
- Cecchetelli, A. (2020, 16. April). Finding nucleic acids with SHERLOCK and DETECTR. Addgene. https://blog.addgene.org/finding-nucleic-acids-with-sherlock-and-detectr ↩
- Centers for Disease Control and Prevention Animal and Plant Health Inspection Service. (2020, 10. September). Bioterrorism: A Brief History. https://www.selectagents.gov/overview/history.htm ↩
- Lewis, G., Jordan, J. L., Relman, D. A., Koblentz, G. D., Leung, J., Dafoe, A., Nelson, C., Epstein, G. L., Katz, R., Montague, M., Alley, E. C., Filone, C. M., Luby, S., Church, G. M., Millett, P., Esvelt, K. M., Cameron, E. E., & Inglesby, T. V. (2020, 8. Dezember). The biosecurity benefits of genetic engineering attribution. Nature Communications, 11(1), 6294. https://doi.org/10.1038/s41467-020-19149-2 ↩
- Alley, E. C., Turpin, M., Liu, A. B., Kulp-McDowall, T., Swett, J., Edison, R., Von Stetina, S. E., Church, G. M., & Esvelt, K. M. (2020). A machine learning toolkit for genetic engineering attribution to facilitate biosecurity. Nature Communications, 11(1), 6293. https://doi.org/10.1038/s41467-020-19612-0 ↩
- Kamens, J. (2015, 28. Januar). The Addgene repository: An international nonprofit plasmid and data resource. Nucleic Acids Research, 43(D1), D1152–D1157. https://doi.org/10.1093/nar/gku893 ↩