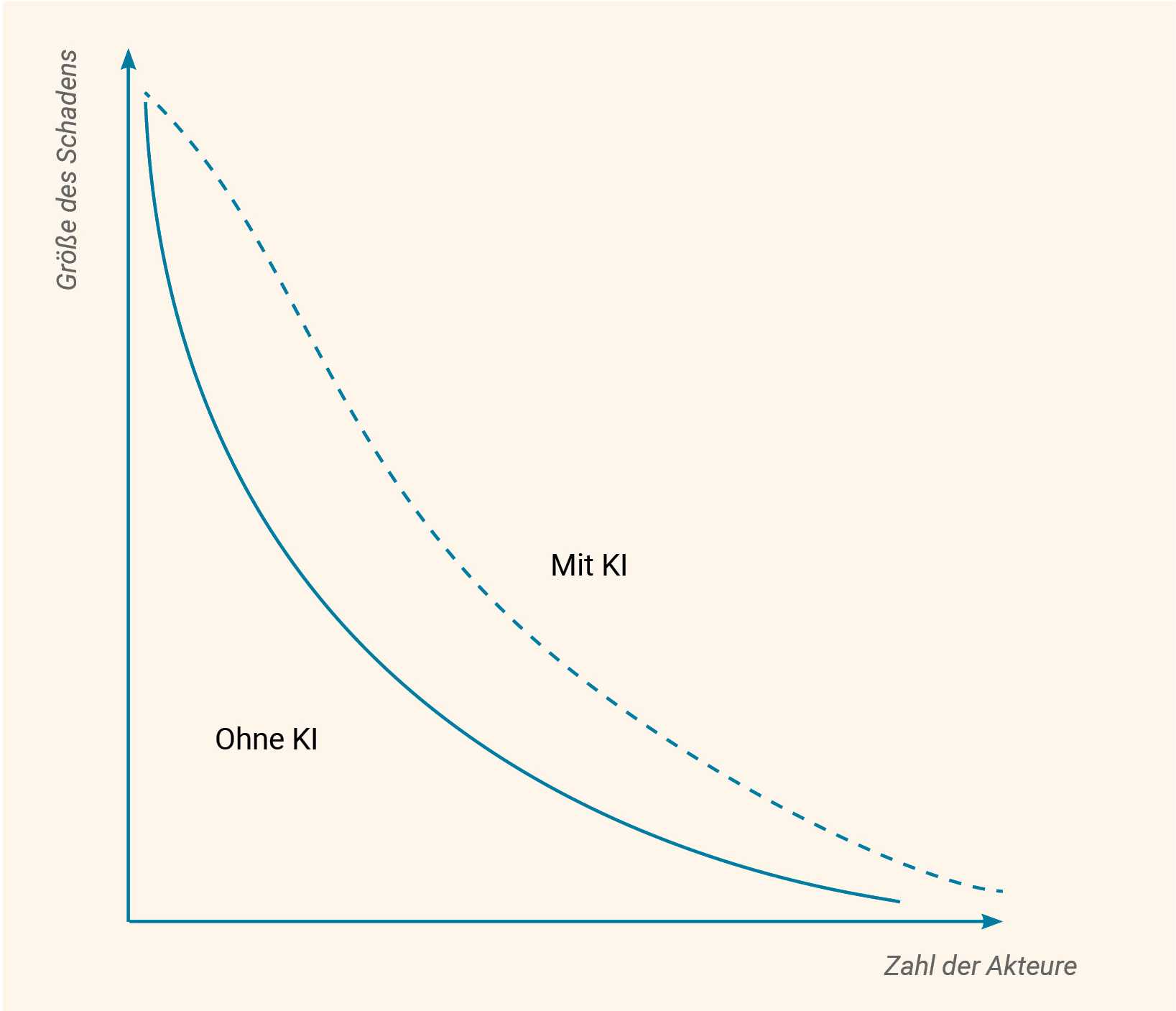
Technologische Implikationen von KI für biologische Risiken
KI wird zunehmend auch in Verbindung mit den Bereichen konventionelle Rüstungskontrolle, Gefechtsfeldmanagement und Datenanalyse diskutiert, wie beispielsweise im Kontext der Ukraine und in Gaza zu sehen ist. Gleichzeitig unterstreicht der wachsende Einfluss von KI in verschiedenen Bereichen die Notwendigkeit, auch über Politik und Governance zu diskutieren. In diesem Kapitel sollen die möglichen Auswirkungen der Konvergenz zwischen Biowissenschaften und KI untersucht werden. Zunächst werden die zahlreichen Vorteile betrachtet, die sich an dieser Schnittstelle ergeben können (mit Beispielen aus den Bereichen Bioabwehr und Biowaffen/Attribution von gentechnischen Veränderungen), um dann die damit verbundenen Risiken zu analysieren und Vorschläge zu ihrer Eindämmung zu entwickeln. Obwohl einige dieser Risiken für die Biologie und die Chemie verallgemeinert werden können, z. B. die Verwendung von KI für das Design von Molekülen/Sequenzen, konzentriert sich dieses Kapitel weitgehend auf biologische Risiken.
Vorteile der Konvergenz von KI und Biowissenschaften
Die jüngsten Fortschritte auf dem Gebiet der Künstlichen Intelligenz (KI) bieten den modernen Biowissenschaften und dem Bioengineering enorme Vorteile. KI kann die rasche Entwicklung von Impfstoffen und Therapeutika unterstützen, einen Beitrag zur Materialwissenschaft leisten, die Wirtschaft ankurbeln und zur Bekämpfung des Klimawandels beitragen.1
KI in der Pandemievorsorge
Die Verbesserung der Pandemievorsorge und -reaktion erfordert auch den Einsatz fortschrittlicher Technologien wie KI, um die Meldung und Analyse von Ausbrüchen zu optimieren.2 Durch die effiziente Verarbeitung von Daten aus Ausbruchsmeldungen und die Analyse von Informationen über zurückkehrende Reisende können KI-Werkzeuge die Häufigkeit der Einschleppung von Infektionskrankheiten modellieren und deren Ursprung zurückverfolgen. Das Europäische Zentrum für die Prävention und die Kontrolle von Krankheiten stellt beispielsweise Werkzeuge für die Analyse von Überwachungsdaten zu Infektionskrankheiten bereit.3 Routinemäßige Überwachungsdaten werden einer statistischen Analyse unterzogen, um Schwankungen in der Krankheitsinzidenz zu erkennen, so dass potenzielle Ausbrüche frühzeitig erkannt und untersucht werden können. In Kombination mit der Genomsequenzierung wird dieser Ansatz entscheidend sein, um Ausbrüche festzustellen und persistente Erregerreservoire aufzudecken.
Darüber hinaus spielt die KI eine Schlüsselrolle bei der Vorhersage des Risikos, das von (neuen) Erregerstämmen ausgeht.4 Schnelle DNA-Analysen, die durch KI-Tools erleichtert werden, ermöglichen es Wissenschaftler*innen, potenzielle Pandemieerreger und Hochrisikovarianten zu identifizieren, bevor sie sich zu weit verbreiteten Ausbrüchen entwickeln.
KI in der Überwachung von Krankheitserregern
Metagenomische Überwachung, wie z. B. die Überwachung von Abwässern, kann Krankheitserreger identifizieren, die in Gemeinschaften zirkulieren.5 Angesichts der enormen Datenmengen, die erzeugt werden, können KI-Werkzeuge wie die Erkennung von Anomalien helfen, (neue) Bedrohungen zu identifizieren.6 bieten eine kostengünstige Alternative zur DNA-Sequenzierung für die kontinuierliche Überwachung von Infektionskrankheiten.7 Die Entwicklung von Panels, die zahlreiche Krankheitserreger nachweisen können, kann komplex sein, aber KI-Optimierungstechniken können diesen Prozess vereinfachen.
KI in der Gentechnik-Attribution
Jüngste Fortschritte zeigen, dass KI-Werkzeuge gentechnisch veränderte Organismen effektiv identifizieren und ihrem Ursprungslabor zuordnen können. Die Designentscheidungen eines Bioingenieurs – z. B. die Wahl des Promotors (Transkriptionsstartpunkt einer DNA-Sequenz), die Optimierung des Codes, die Auswahl funktioneller Gene und die Wahl der Klonierungsmethode – bilden zusammen die „Signatur“ dieses Designers.8 Mehrere neue Techniken wurden entwickelt, um diese Signaturen zu erfassen. Ihre Analyse erfordert eine komplexe Datenanalyse, die durch und KI-Werkzeuge verbessert werden könnte. Diese Werkzeuge sind zwar vielversprechend bei der Identifizierung von Akteuren, die biologische Gefahrenstoffe entwickeln, ihre Wirksamkeit bei der Attribution kann jedoch beeinträchtigt werden, wenn die Akteure Designentscheidungen manipulieren können, um der Entdeckung zu entgehen.
KI in der Synthetischen Biologie
Die Synthetische Biologie bietet ein großes Potenzial zur Bewältigung wichtiger gesellschaftlicher Herausforderungen. Ein Haupthindernis ist jedoch, dass biologische Systeme derzeit nicht so genau vorhergesagt werden können wie physikalische oder chemische Systeme, die auch simuliert werden können. Diese Einschränkung hat sowohl praktische als auch grundlegende Konsequenzen. In der Praxis können biologische Systeme (z. B. Proteine, Stoffwechselwege, Zellen) nicht nach bestimmten Anforderungen (z. B. Bindungsaffinität, Produktionsraten) entworfen werden. Grundsätzlich fehlt ein tiefes Verständnis der Mechanismen, die beobachtbare Merkmale oder Eigenschaften von Organismen hervorbringen. KI und maschinelles Lernen sind vielversprechende Methoden, um die in der Synthetischen Biologie benötigte Vorhersagekraft zu verbessern, und können während des gesamten Prozesses der Synthetischen Biologie eingesetzt werden.
In den letzten zwei Jahrzehnten wurden beispielsweise verschiedene Werkzeuge für maschinelles Lernen entwickelt, die das Enzym-Engineering unterstützen, indem sie Ansätze vereinfachen und den erforderlichen Screening-Aufwand verringern. ML kann Informationen über Enzymsequenzen und -eigenschaften verarbeiten und daraus neue Informationen ableiten, die diese Eigenschaften wahrscheinlich verbessern oder verfeinern. Diese Algorithmen haben zahlreiche Anwendungen in der Synthetischen Biologie, einschließlich der Optimierung von genetischen und metabolischen Netzwerken, der Steuerung der Enzymevolution, der Vorhersage der kinetischen Eigenschaften von nicht charakterisierten Enzymen und sogar das Neuerschaffen von ganzen Proteinen.
Risiken der KI-Bio-Konvergenz
Expert*innen, die die potenziellen Risiken von KI-Methoden für die Biowissenschaften analysieren, konzentrieren sich derzeit auf zwei Hauptfragen.9 Erstens: Verringern die Fortschritte bei den KI-Werkzeugen die Hürden für die Entwicklung von Krankheitserregern? Einige Expert*innen vermuten, dass dies der Fall ist.10 Zweitens: Welche KI-Modelle und -Werkzeuge bergen das größte Missbrauchsrisiko?
KI-Werkzeuge, die ein potenzielles Sicherheitsrisiko darstellen, lassen sich in drei Kategorien einteilen:11 Large Language Models (LLMs), Biodesign-Tools (BDTs) und KI-gestützte Automatisierung in den Biowissenschaften. Obwohl diese Kategorien bei der Bewertung der jeweiligen potenziellen Biosicherheitsrisiken hilfreich sein können, verschwimmen die Grenzen zwischen diesen Fähigkeitstypen zunehmend.12
Large Language Models (LLMs)
Obwohl in natürlicher Sprache nicht speziell für die Förderung der Biowissenschaften entwickelt wurden, werden sie die biowissenschaftliche Forschung erheblich beeinflussen, indem sie Ausbildung, Grundlagenforschung und Laborkapazitäten unterstützen. LLMs sind in der Lage, Informationen aus einer Vielzahl von Quellen schnell zusammenzuführen und in verständlicher Sprache darzustellen. Im Grunde stellt dies bereits ein Dual-Use-Problem dar, da LLMs Wissen für die legitime Wissenschaft bereitstellen, aber auch für illegitime Zwecke genutzt werden können.13 Gegenwärtig scheinen diese Modelle vor allem für Nutzer*innen mit einem gewissen Grundwissen von Vorteil zu sein. Aber mehrere Expert*innen sind der Ansicht, dass sie auch Personen, die mit einem Thema gänzlich unvertraut sind, schnell zu einem Verständnis verhelfen können, das mit dem von Studierenden oder sogar Promovierenden vergleichbar ist. Laut einem Bericht der NTI (Nuclear Threat Initiative)14 teilen einige Expert*innen die Einschätzung, dass es den Instrumenten an Präzision und Genauigkeit mangelt, wenn es um technisch komplexere Fragen geht. Sie sind der Ansicht, dass die derzeitigen LLMs wahrscheinlich keine wesentlichen neuen Erkenntnisse über biologische Systeme liefern werden. Einige LLMs sind speziell auf wissenschaftliche Literatur ausgerichtet und könnten für Studierende und andere Personen, die sich mit technischen Themen vertraut machen wollen, nützlicher sein.
Es wurden Versuche unternommen, die damit verbundenen Risiken zu bewerten.15 Diese deuten darauf hin, dass die derzeitigen LLMs das biologische Risiko nicht signifikant erhöhen. Allerdings gab es auch erhebliche Kritik am Studiendesign und den verwendeten statistischen Methoden.16 So wurde unter anderem darauf hingewiesen, dass diese Berichte unter einer asymmetrischen Risikobewertung leiden könnten, bei der nur negative Ergebnisse veröffentlicht werden (da positive Ergebnisse, die zeigen, welchen potenziellen Schaden diese Modelle anrichten könnten, als gefährlich angesehen werden könnten). Diese Diskrepanz könnte möglicherweise die Wahrnehmung des tatsächlichen Risikos verzerren.
LLMs könnten potenziell dabei helfen, auf der Grundlage veröffentlichter Informationen molekularbiologische Experimente zu planen und Fehler darin zu suchen sowie Roboterplattformen zur Durchführung von Experimenten zu programmieren. Diese Fähigkeiten können es weniger notwendig machen, dass Einzelpersonen die technischen Fähigkeiten entwickeln, die für die Durchführung der Experimente selbst erforderlich sind. Während die derzeitigen LLMs in der Regel auf textbasierte Informationen beschränkt sind, können einige Modelle auch Informationen aus Bildern und Videos einbeziehen, wodurch sie möglicherweise in der Lage sind, effektiveres Feedback und Vorschläge zur Fehlerbehebung bei Labortechniken in Echtzeit zu liefern. Diese KI-gestützten Laborassistenten könnten viele nützliche Anwendungen zur Unterstützung von Forschenden bieten, insbesondere solchen, die am Anfang ihrer Karriere stehen. Dieselben KI-gestützten Assistenten könnten jedoch auch die Laborarbeit zu unlauteren Zwecken unterstützen.
Biologische Design-Tools (BDTs)
Die zweite Kategorie von KI-Tools mit einem potenziellen Missbrauchsrisiko umfasst . Es wird angenommen, dass Large Language Models (LLMs), die auf biologischen Daten basieren, die effektivsten BDTs sein könnten. LLMs können mit DNA- und RNA-Sequenzierungsmustern trainiert werden, um Modifikationen und Regulierungsmechanismen innerhalb dieser Sequenzen vorherzusagen. Darüber hinaus können LLMs, die auf Proteindaten trainiert wurden, für verschiedene Anwendungen eingesetzt werden, z. B. zur Vorhersage von Proteinstrukturen, zur Generierung neuer Proteine, zur Annotation von Proteinfunktionen und zur Vorhersage von Proteininteraktionen. Derzeit werden BDTs häufig der Öffentlichkeit zugänglich gemacht, unabhängig davon, ob sie von der Wissenschaft (z. B. RFDiffusion) oder der Industrie (z. B. ProGen2) entwickelt wurden. Während die heutigen BDTs auf die Erzeugung von Proteinen mit relativ einfachen Einzelfunktionen beschränkt sind, dürften künftige Iterationen in der Lage sein, Proteine17 und Enzyme mit mehreren Funktionen zu optimieren. Neben Werkzeugen für das Design von Proteinen oder Organismen gibt es auch andere maschinelle Lernwerkzeuge mit doppeltem Verwendungszweck, darunter solche, die Eigenschaften wie die Immunabwehr vorhersagen oder das funktionelle Verständnis des menschlichen Genoms fördern. Deep-Learning-Architekturen wie Enformer verbessern die Genauigkeit von Vorhersagen der Genexpression aus DNA-Sequenzen erheblich.18
Mehrere Expert*innen befürchten, dass KI-Werkzeuge für das Proteindesign es den DNA-Anbietern erschweren könnten, die bestellte DNA umfassend zu überwachen. Da die derzeitigen DNA-Screening-Methoden auf einem Vergleich der in Auftrag gegebenen DNA mit der DNA eines Krankheitserregers beruhen, könnten sich die neu entworfenen Proteine dieser Kontrolle entziehen. Diese Werkzeuge könnten so die Schaffung neuer Bedrohungen unterstützen (um Toxin- oder Krankheitserregerdesigns zu erzeugen, die in der Natur nicht vorkommen) und sich dem DNA-Sequenzierungsscreening entziehen.19
KI-gestützte Automatisierung der Biowissenschaften
Vereinfacht ausgedrückt wird unter KI-gestützter automatisierter Wissenschaft die Übertragung eines, mehrerer oder aller Schritte des wissenschaftlichen Prozesses auf KI verstanden. Dem NTI-Bericht zufolge gab es zum Zeitpunkt seiner Erstellung mehrere Beispiele für Forschungsprojekte, bei denen mindestens ein Schritt des wissenschaftlichen Prozesses automatisiert wurde. Zu diesen Schritten gehören die anfängliche wissenschaftliche Literaturrecherche, die Entwicklung von Hypothesen, die Planung und Durchführung von Experimenten mithilfe von Robotern, die Analyse der Ergebnisse und die Anpassung der Hypothese. Jede dieser Fertigkeiten könnte den wissenschaftlichen Prozess erheblich beschleunigen, indem sie Möglichkeiten zur Skalierung bietet, die Zahl der erforderlichen Experimente verringert und von Menschen verursachte Verzögerungen und Fehler reduziert. Die Möglichkeit einer vollständigen Automatisierung biologischer Experimente wird noch diskutiert.
Ein neuerer Ansatz zur Automatisierung der Wissenschaft besteht in der Entwicklung autonomer , die mit mehreren KI-Werkzeugen (z. B. LLMs) interagieren können, um komplexe Aufgaben zu koordinieren und auszuführen. Ein Beispiel aus der Chemie ist ChemCrow,20 ein LLM-basierter Agent, der entwickelt wurde, um Aufgaben in den Bereichen organische Synthese, Arzneimittelentdeckung und Materialdesign auszuführen. Diese Art von Werkzeugen kann verwendet werden, um das Internet nach relevanten Informationen zu durchsuchen, sich über Anwendungsprogrammierschnittstellen (APIs) mit der Robotik zu verbinden und sich sogar mit anderen LLMs zu verbinden, um Aufgaben zu delegieren, wie das Ibuprofen-Syntheseprojekt21 gezeigt hat.
KI in der Arzneimittelentwicklung
Derzeit besteht die Notwendigkeit, die Kosten und den Zeitaufwand für die Entdeckung von Arzneimitteln zu senken. Dies wird die Entwicklung von Behandlungsmöglichkeiten für Krankheiten erleichtern, für die weniger Forschungsmittel zur Verfügung stehen, wie z. B. Tropenkrankheiten und vernachlässigte Krankheiten, und die Verfügbarkeit neuer Arzneimittel insgesamt erhöhen. Wissenschaftler*innen machen Fortschritte mit Wissenschaftsrobotern wie Eve,22 einem System, das die Entdeckung von Arzneimitteln in einem frühen Stadium automatisiert, um neue Behandlungen für vernachlässigte tropische Krankheiten zu finden. In den letzten Jahren wurde auch zunehmend diskutiert, wie KI-Technologien für die Arzneimittelentdeckung möglicherweise missbraucht werden könnten.23
Risikominderung und Governance
Expert*innen haben mehrere Methoden vorgeschlagen, um die Risiken von LLMs und BDTs anzugehen. Eine davon ist die Bewertung der Modelle vor ihrer Freigabe. Die Charakterisierung von Risiken und die Bewertung der Wirksamkeit von Schutzmaßnahmen vor der Freigabe würde für Entwickler*innen einen Anreiz darstellen, schädliches Modellverhalten während der gesamten Trainings- und Nutzungsphase zu bereinigen.
Eine andere Methode, die Kompromisse in Betracht zieht, ist die Abwägung zwischen der Notwendigkeit eines uneingeschränkten Zugangs und Sicherheitsbedenken. Verschiedene Interessengruppen müssen Beweise sammeln und Werkzeuge entwickeln, um zu beurteilen, wann die Vorteile der Freigabe eines Modells durch die Risiken aufgewogen werden und dabei vielleicht auch Schritte zur Benutzerauthentifizierung für leistungsfähigere Modelle in Betracht ziehen.
Die Charakterisierung von Risiken und die Bewertung der Wirksamkeit von Schutzmaßnahmen vor der Freigabe würde für Entwickler*innen einen Anreiz darstellen, schädliches Modellverhalten während der gesamten Trainings- und Nutzungsphase zu bereinigen.
Expert*innen empfehlen außerdem, die Biosicherheitsmaßnahmen an der Schnittstelle zwischen digitaler und physischer Welt zu verstärken. Da der Zugang zu synthetischer DNA für die Umwandlung biologischer Designs in physische Wirkstoffe von entscheidender Bedeutung ist, haben führende Unternehmen der Branche freiwillig damit begonnen, Gensyntheseaufträge zu überprüfen (indem sie die Identität der Kunden verifizieren und die Bestellungen überwachen, um sicherzustellen, dass Sequenzen, die Schaden anrichten könnten, nicht unangemessen freigegeben werden). Sie setzen sich außerdem für eine gesetzliche Grundlage ein. Die in den USA vom Weißen Haus im Oktober 2023 erlassene KI-Durchführungsverordnung (Executive Order 14110) war ein Schritt in diese Richtung, da sie die Verwendung von kontrollierter DNA in der staatlich finanzierten Forschung vorschreibt. Die EU hat jedoch noch keine Regelungen zu diesem Thema erlassen. Im Jahr 2024 wurde eine globale Grundlage für das Screening von Nukleinsäuresynthese vorgeschlagen, der sogenannte „Common Mechanism for DNA Synthesis Screening“.24 Dieses Instrument wurde in Zusammenarbeit mit einem technischen Konsortium von Expert*innen aus den Bereichen DNA-Synthese, synthetische Biologie, Biosicherheit und Politik entwickelt, um bestehende Hindernisse zu überwinden. Die Common-Mechanism-Software verwendet mehrere Methoden, um bedenkliche Sequenzen zu identifizieren, sie taxonomisch mit regulierten Krankheitserregern abzugleichen und gutartige Gene für die Synthese freizugeben.
Ende 2023 wurden in den USA mehrere neue Anforderungen an leistungsstarke KIxBio-Modelle eingeführt. Die Executive Order schreibt umfassende Maßnahmen vor, um die von KI-Systemen ausgehenden nationalen Sicherheitsrisiken zu mindern. Unternehmen, die „Dual-Use-Basismodelle“ entwickeln, müssen regelmäßig über ihre Modellentwicklungspläne und Risikomanagementstrategien berichten, einschließlich der Absicherung von Modellgewichten und der Durchführung von zur Identifizierung gefährlicher Fähigkeiten. Für KI-Modelle, die mit biologischen Sequenzdaten trainiert werden, gelten neue Berichts- und Compliance-Anforderungen.
- Carter, S. R., Wheeler, N., Chwalek, S., Isaac, C., & Yassif, J. M. (2023). The Convergence of Artificial Intelligence and the Life Sciences: Safeguarding Technology, Rethinking Governance, and Preventing Catastrophe. Nuclear Threat Initiative. https://www.nti.org/analysis/articles/the-convergence-of-artificial-intelligence-and-the-life-sciences/ ↩
- Syrowatka, A., Kuznetsova, M., Alsubai, A., Beckman, A. L., Bain, P. A., Craig, K. J. T., Hu, J., Jackson, G. P., Rhee, K., & Bates, D. W. (2021, 10. Juni). Leveraging artificial intelligence for pandemic preparedness and response: A scoping review to identify key use cases. Npj Digital Medicine, 4(1), 96. https://doi.org/10.1038/s41746-021-00459-8 ↩
- European Centre for Disease Prevention and Control. (2018, 17. Dezember). New tools for public health experts: Outbreak detection and epidemiological reports. Europäische Union, European Centre for Disease Prevention and Control (ECDC). https://www.ecdc.europa.eu/en/news-events/new-tools-public-health-experts-outbreak-detection-and-epidemiological-reports ↩
- Njage, P. M. K., Henri, C., Leekitcharoenphon, P., Mistou, M., Hendriksen, R. S., & Hald, T. (2018). Machine Learning Methods as a Tool for Predicting Risk of Illness Applying Next‐Generation Sequencing Data. Risk Analysis, 39(6), 1397–1413. https://doi.org/10.1111/risa.13239; Zhao, A. P., Li, S., Cao, Z., Hu, P. J.-H., Wang, J., Xiang, Y., Xie, D., & Lu, X. (2024, 2. Juni). AI for science: Predicting infectious diseases. Journal of Safety Science and Resilience, 5(2), 130–146. https://doi.org/10.1016/j.jnlssr.2024.02.002 ↩
- Ahmed, W., Angel, N., Edson, J., Bibby, K., Bivins, A., O’Brien, J. W., Choi, P. M., Kitajima, M., Simpson, S. L., Li, J., Tscharke, B., Verhagen, R., Smith, W. J. M., Zaugg, J., Dierens, L., Hugenholtz, P., Thomas, K. V., & Mueller, J. F. (2020, 1. August). First confirmed detection of SARS-CoV-2 in untreated wastewater in Australia: A proof of concept for the wastewater surveillance of COVID-19 in the community. Science of The Total Environment, 728, 138764. https://doi.org/10.1016/j.scitotenv.2020.138764 ↩
- Eze, P. U., Geard, N., Mueller, I., & Chades, I. (2023). Anomaly Detection in Endemic Disease Surveillance Data Using Machine Learning Techniques. Healthcare, 11(13), 1896. https://doi.org/10.3390/healthcare11131896; Ren, H., Li, Y., & Huang, T. (2023). Anomaly Detection Models for SARS-CoV-2 Surveillance Based on Genome k-mers. Microorganisms, 11(11), 2773. https://doi.org/10.3390/microorganisms11112773 ↩
- Metsky, H. C., Welch, N. L., Pillai, P. P., Haradhvala, N. J., Rumker, L., Mantena, S., Zhang, Y. B., Yang, D. K., Ackerman, C. M., Weller, J., Blainey, P. C., Myhrvold, C., Mitzenmacher, M., & Sabeti, P. C. (2022, 3. März). Designing sensitive viral diagnostics with machine learning. Nature Biotechnology, 40(7), 1123–1131. https://doi.org/10.1038/s41587-022-01213-5 ↩
- Alley, E. C., Turpin, M., Liu, A. B., Kulp-McDowall, T., Swett, J., Edison, R., Von Stetina, S. E., Church, G. M., & Esvelt, K. M. (2020, 8. Dezember). A machine learning toolkit for genetic engineering attribution to facilitate biosecurity. Nature Communications, 11(1), 6293. https://doi.org/10.1038/s41467-020-19612-0 ↩
- Carter et al., 2023. ↩
- Sandbrink, J. B. (2023). Artificial intelligence and biological misuse: Differentiating risks of language models and biological design tools (Version 8). arXiv. https://doi.org/10.48550/ARXIV.2306.13952 ↩
- Rose, S., & Nelson, C. (2023, 18. Oktober). Understanding AI-Facilitated Biological Weapon Development. The Centre for Long-Term Resilience. https://www.longtermresilience.org/post/report-launch-examining-risks-at-the-intersection-of-ai-and-bio ↩
- Toews, R. (2023, 16. Juli). The Next Frontier For Large Language Models Is Biology. Forbes. https://www.forbes.com/sites/robtoews/2023/07/16/the-next-frontier-for-large-language-models-is-biology/ ↩
- Sandbrink, 2023. ↩
- Carter et al., 2023. ↩
- Rose et al., 2023; Mouton, C., Lucas, C., & Guest, E. (2024, 25. Januar). The Operational Risks of AI in Large-Scale Biological Attacks: Results of a Red- Team Study. (RAND). ↩
- Marcus, G. (2024, 4. Februar). When looked at carefully, OpenAI’s new study on GPT-4 and bioweapons is deeply worrisome. Marcus on AI. https://garymarcus.substack.com/p/when-looked-at-carefully-openais ↩
- Spotlight on protein structure design. (2024, 15. Februar). Nature Biotechnology, 42(2), 157–157. https://doi.org/10.1038/s41587-024-02150-1 ↩
- Avsec, Ž., Agarwal, V., Visentin, D., Ledsam, J. R., Grabska-Barwinska, A., Taylor, K. R., Assael, Y., Jumper, J., Kohli, P., & Kelley, D. R. (2021). Effective gene expression prediction from sequence by integrating long-range interactions. https://doi.org/10.1101/2021.04.07.438649 ↩
- Carter et al., 2023. ↩
- Bran, A. M., Cox, S., Schilter, O., Baldassari, C., White, A. D., & Schwaller, P. (2024, 8. Mai). Augmenting large language models with chemistry tools. Nature Machine Intelligence, 6(5), 525–535. https://doi.org/10.1038/s42256-024-00832-8 ↩
- Boiko, D. A., MacKnight, R., & Gomes, G. (2023, 11. April). Emergent autonomous scientific research capabilities of large language models (Version 1). arXiv. https://doi.org/10.48550/ARXIV.2304.05332 ↩
- Williams, K., Bilsland, E., Sparkes, A., Aubrey, W., Young, M., Soldatova, L. N., De Grave, K., Ramon, J., De Clare, M., Sirawaraporn, W., Oliver, S. G., & King, R. D. (2015, 6. März). Cheaper faster drug development validated by the repositioning of drugs against neglected tropical diseases. Journal of The Royal Society Interface, 12(104), 20141289. https://doi.org/10.1098/rsif.2014.1289 ↩
- Urbina, F., Lentzos, F., Invernizzi, C., & Ekins, S. (2022, 7. März). Dual use of artificial-intelligence-powered drug discovery. Nature Machine Intelligence, 4(3), 189–191. https://doi.org/10.1038/s42256-022-00465-9 ↩
- Wheeler, N. E., Carter, S. R., Alexanian, T., Isaac, C., Yassif, J., & Millet, P. (2024, 20. Juni). Developing a Common Global Baseline for Nucleic Acid Synthesis Screening. Applied Biosafety, 29(2), 71–78. https://doi.org/10.1089/apb.2023.0034 ↩