KI im Labor: Die Zukunft des Biodesigns
In diesem Kapitel wird betrachtet, wie KI derzeit das Biodesign revolutioniert, wobei neue Werkzeuge Fortschritte in der Proteintechnik und der Genforschung ermöglichen. Es beleuchtet die jüngsten Entwicklungen, die die wissenschaftlichen Möglichkeiten erweitern und gleichzeitig Sicherheitsbedenken hervorrufen, einschließlich der möglichen Entwicklung von schädlichen biologischen Agenzien und Cyber-Bio-Bedrohungen. Das Kapitel erörtert die derzeitigen Bemühungen, diese Risiken durch technische Sicherheitsvorkehrungen, kontrollierten Zugang und Wasserzeichen zu beobachten und zu verringern, und es unterstreicht die Notwendigkeit einer abgestimmten internationalen Politik, die sichere Innovationen anleiten kann.
Im letztjährigen CNTR Monitor1 wurden Beispiele für das Zusammenspiel von KI und Biowissenschaften behandelt. KI wird zunehmend in der Bioüberwachung eingesetzt, um dabei zu helfen, ungewöhnliche Muster in Gesundheitsdaten, Erregersequenzen oder Umweltsignalen zu erkennen, die auf Ausbrüche von Infektionskrankheiten oder biologische Bedrohungen hinweisen könnten. Gleichzeitig werden neue Methoden entwickelt, um gentechnisch veränderte Organismen zu identifizieren und zu ihrem Ursprungslabor zurückzuverfolgen. Diese Fortschritte eröffnen zwar vielversprechende Möglichkeiten, bringen aber auch neue Risiken mit sich, die sorgfältig eingehegt werden müssen.
KI-Protein-Werkzeuge
Biodesigninstrumente (englisch „biological design tools“, BDTs) werden auf biologischen Datensätzen trainiert, im Gegensatz zu großen Sprachmodellen (LLMs), die auf natürlicher menschlicher Sprache trainiert werden. Anders als LLMs werden die meisten Biodesigninstrumente von Forschenden in den Lebenswissenschaften oder der Industrie für spezielle Anwendungen und nicht für den öffentlichen Gebrauch entwickelt. Ihre Bedeutung wird jedoch zunehmend anerkannt, wie die Verleihung des Nobelpreises für Chemie im Jahr 2024 unterstreicht. Mit dem Preis wurden David Baker für das computergestützte Proteindesign sowie Demis Hassabis und John Jumper von Google für AlphaFold geehrt – letzteres ist ein Programm, das das alte Problem der Vorhersage von 3D-Strukturen von Proteinen aus Aminosäuresequenzen gelöst hat. Das Feld entwickelt sich rasant weiter, mit Durchbrüchen wie dem erwähnten AlphaFold von DeepMind für die Vorhersage von Proteinstrukturen und AlphaProteo, einer 2024 veröffentlichten Software für das Proteindesign. Es gibt ferner Rosetta Commons, das von David Baker und seiner Gruppe entwickelt wurde, mit seinen vielfältigen Anwendungen und Tools wie RFdiffusion, das KI zur Entwicklung neuartiger Proteinstrukturen einsetzt, indem es vorhersagt, wie sich Aminosäurenketten zu dreidimensionalen Formen auffalten, und ESM3 von EvolutionaryScale, einem Foundation Model für die Lebenswissenschaften, das auf DNA-, RNA- und Aminosäuresequenzen trainiert wurde.
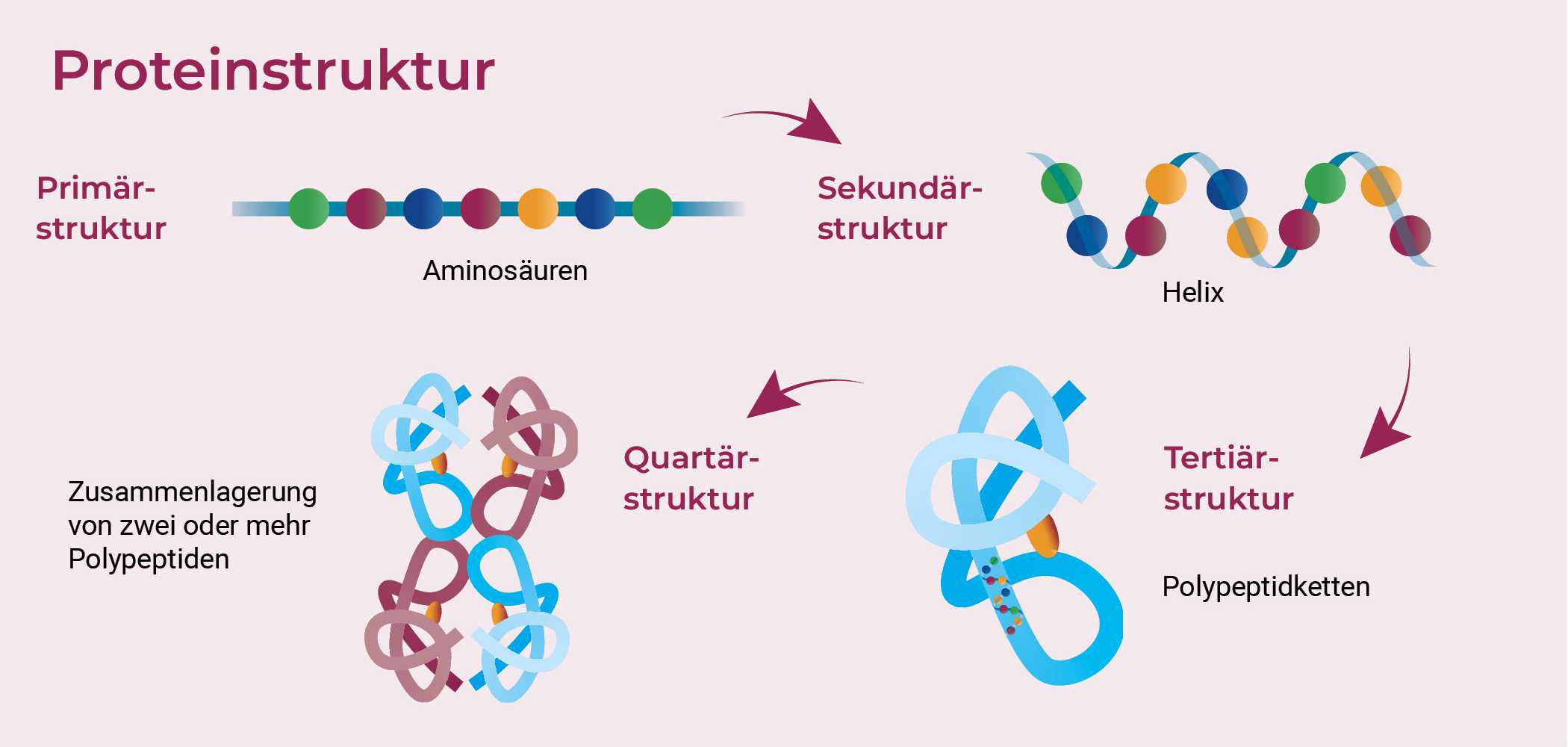
Vorhersage der Proteinfaltung in großem Maßstab
In der Zelle werden Proteine als lineare Ketten synthetisiert, die sich von selbst in bestimmte Formen falten, die ihre Funktionen bestimmen. Unter normalen Bedingungen faltet sich eine bestimmte Sequenz in eine Hauptform, wobei einige Proteine auch mehrere Formen annehmen können, was zu unterschiedlichen Funktionen oder sogar Krankheiten führen kann. Die Form ist entscheidend, denn sie steuert die Interaktion des Proteins mit anderen Molekülen. Wissenschaftler*innen versuchen, die Struktur eines Proteins anhand seiner Sequenz mit Hilfe von Computern vorherzusagen oder Sequenzen zu entwerfen, die sich in die gewünschte Form auffalten. Experimentelle Methoden wie , und sind nach wie vor kostspielig. Im Gegensatz dazu ist die Bestimmung von Proteinsequenzen aus Genomdaten wesentlich kostengünstiger geworden, was zu Milliarden von Proteinsequenzen, aber nur einigen hunderttausend experimentell aufgeklärten Strukturen geführt hat.2 Das ultimative Ziel ist es, Proteine mit vorgegebenen Strukturen zu entwerfen. Um den Fortschritt bei der Vorhersage von Proteinstrukturen zu bewerten und Innovationen voranzutreiben, wurde das Critical Assessment of Structure Prediction (CASP) ins Leben gerufen, das seit 1994 weltweit durchgeführt wird. CASP ist ein Wettbewerb, bei dem Forschungsteams versuchen, die 3D-Formen von Proteinen anhand ihrer Aminosäuresequenzen vorherzusagen und zu testen, wie nahe ihre Vorhersagen an experimentell ermittelten Strukturen liegen. Im Jahr 2018 erregte CASP13 Aufmerksamkeit, als das KI-Programm AlphaFold von DeepMind gewann. Ein verbessertes AlphaFold2 dominierte dann CASP14 im Jahr 2020 und erreichte mit Hilfe von neuronalen Netzen eine Genauigkeit von etwa 90% bei mäßig schwierigen Proteinzielen.
Über Proteine hinaus
KI hilft auch bei der Entdeckung von , verbessert die Diagnostik und optimiert die Bioproduktion. In anderen Wissenschaften kann die KI Experimente autonom steuern. Diese Fortschritte bergen jedoch auch Sicherheitsrisiken, da sie zur Entwicklung von Toxinen, zur Verschärfung von Krankheitserregern und zur Steigerung anderer biologischer Bedrohungen missbraucht werden könnten. Obwohl Fortschritte bei den Werkzeugen für das Proteindesign und anderen Biodesigninstrumenten Risiken hervorrufen können, wurden bisher nur wenige Sicherheitsvorkehrungen getroffen. Während erhebliche Anstrengungen und Investitionen in LLM-Sicherheitsvorkehrungen geflossen sind, erfahren Biodesigninstrumente vergleichsweise wenig Aufmerksamkeit. Bei ausreichendem Zugang ist es jedoch möglich, dass jemand absichtlich oder versehentlich schädliche Krankheitserreger oder Toxine erzeugt.
Einer der ausführlichsten Berichte zu diesem Thema stammt von NTI-Bio;3 darin werden drei Schlüsselfragen untersucht:
- Welche eingebauten Sicherheitsvorkehrungen könnten eingerichtet werden?
- Wie sollte der Zugang zu diesen Tools verwaltet werden (Abwägung zwischen Open-Source- und vollständig kontrollierten Modellen)?
- Wie können wir Lösungen für die Biosicherheit entwickeln, die Risiken mindern und gleichzeitig das volle Potenzial dieser Technologien zum Nutzen der Gesellschaft bewahren?
Die Autor*innen des Berichts sprachen mit Expert*innen, organisierten Workshops und führten Peer Reviews durch, um mögliche Sicherheitsvorkehrungen für Modellentwickler*innen und die breitere Biosicherheitsgemeinde zu identifizieren. Sie diskutierten mehrere Ansätze, z. B. die Kontrolle des Zugriffs bereits auf der Ebene der Trainingsdaten und in der Phase der Datenerfassung. Weitere wichtige Punkte waren die Regelung des Zugangs zu Computerressourcen und verantwortungsbewusste Ausbildungsmethoden. Vor der Veröffentlichung könnten eingebaute Sicherheitsvorkehrungen und Modellevaluierungen implementiert werden, gefolgt von gesteuertem Zugriff auf das Modell in der letzten Phase, um eine verantwortungsvolle Nutzung zu gewährleisten.
Als Sicherheitsvorkehrungen könnten zum Beispiel bestimmte Arten von Outputs eingeschränkt, Abfragen auf verdächtige Muster überwacht oder Warnsysteme für risikoreiche Anfragen eingerichtet werden. Im Vergleich zur Situation bei auf die Chemie zugeschnittenen Werkzeugen wie ChemCrow könnten Sicherheitsvorkehrungen in biologischen Anwendungen eine größere Herausforderung darstellen, da schädliche biologische Ergebnisse schwieriger zu definieren oder zu erkennen sind. Während ein toxisches Molekül eindeutig identifizierbar ist, kann eine Proteinsequenz oder ein genetisches Design zwar harmlos erscheinen, aber dennoch Gefahrenpotenzial haben, was Screening und Interventionen erschwert.
Ähnlich wie beim Screening von Nukleinsäurebestellungen (im Kapitel über DNA-Synthese behandelt) könnten die Outputs der Modelle überwacht werden, um potenziellen Missbrauch zu erkennen, so dass Anfragen, bei denen es um gefährliche Designs gehen könnte, gekennzeichnet oder direkt abgelehnt werden können. Genau wie das DNA-Synthese-Screening würde dieser Ansatz jedoch die globale Einrichtung einer gemeinsamen Informationsquelle oder Datenbank erfordern, die allerdings selbst eine Gefährdung darstellen könnte, indem sie Wissen verbreiten könnte, das Schaden ermöglicht.
Auf einer anderen Ebene geht es darum, die Benutzer*innen und die Absicht hinter einem biologischen Design zu verstehen und dann entsprechend den Zugriff zu steuern. Der Zugang könnte dabei auf einem Spektrum liegen, das vom vollständigen Zugriff auf das Instrument und den Code bis zum eingeschränkten Zugang reicht, der auf Outputs per begrenzt ist, über welches Benutzer*innen Inputs senden und Outputs empfangen können, ohne das zugrundeliegende Modell sehen oder verändern zu können. Es ist wichtig, ein Gleichgewicht zu finden: Sicherheitsexpert*innen bevorzugen möglicherweise die Kontrolle über das Werkzeug selbst, um zu verhindern, dass Sicherheitsvorkehrungen entfernt werden, während Forschende häufig für Offenheit eintreten, um gleiche Zugänglichkeit, Reproduzierbarkeit und Transparenz zu gewährleisten. Kein einzelnes Land und keine einzelne Region sollte exklusiven Zugang zu Instrumenten haben, die beispielsweise bei der Entwicklung von Impfstoffen eingesetzt werden können.
Große Unternehmen wie Google DeepMind tragen dazu bei, die Risiken zu mindern, die mit den von ihnen entwickelten leistungsstarken Tools verbunden sind.4 Dazu gehört auch die Einschränkung des Zugriffs auf gewisse bedenkliche Sequenzen. Einige Nutzer*innen argumentieren jedoch, dass dies die legitime Forschung einschränke und die Frage aufwerfe, wer bestimmen sollte, was als legitime Wissenschaft zu gelten habe. Sie haben sich auch mit Wasserzeichen in biomolekularen Strukturen beschäftigt, um identifizierbare Marker oder Signaturen in synthetische oder veränderte DNA oder biologische Produkte einzubetten. Dies würde dazu beitragen, deren Herkunft zurückzuverfolgen, Authentizität zu überprüfen, Manipulationen aufzudecken und die Rechenschaftspflicht im Falle eines Missbrauchs oder einer unbeabsichtigten Freisetzung zu unterstützen. Diese Position ist auch auf Kritik gestoßen, denn es wird befürchtet, dass solche Maßnahmen als wettbewerbswidrig angesehen werden könnten, insbesondere wenn sie von einem Unternehmen wie Google umgesetzt werden.
Um einige dieser Probleme zu lösen, könnte ein Wasserzeichen-Regime dieser Art aufgesetzt werden: Wissenschaftler*innen erhalten einen autorisierten privaten Schlüssel, der mit ihrer Identität verknüpft ist und es ihnen ermöglicht, zu von generativen Modellen entworfenen Proteinsequenzen Wasserzeichen hinzuzufügen.5 Ein Programm zur Erkennung von Wasserzeichen kann die Autorisierung lokal überprüfen, ohne Sequenzdaten auf einem Server zu speichern. Diese Wasserzeichen verknüpfen die synthetisierten Sequenzen sicher mit den Forschenden und ermöglichen die Rückverfolgbarkeit. Wenn ein verdächtiges Protein identifiziert wird, können die Behörden seine Herkunft zurückverfolgen, indem sie die Wasserzeichen mit den privaten Schlüsseln abgleichen. Die robusten Wasserzeichen erhöhen die Biosicherheit, indem sie von Missbrauch abschrecken und es gleichzeitig den Forschenden ermöglichen, geistige Eigentumsrechte an ihren Sequenzen geltend zu machen.
Solche Rahmenwerke bieten eine hervorragende technische Grundlage für das Management von mit KI-gestützten Biodesigninstrumenten verbundenen Risiken. Es ist jedoch ebenso wichtig, diese Bemühungen in gemeinsame internationale Regelungsstrukturen einzubetten, anstatt sich allein auf freiwillige Unternehmensmaßnahmen oder einseitige nationale Kontrollen zu verlassen. Wie beim DNASynthese-Screening sollten transparente, kompatible Sicherheitsvorkehrungen angestrebt werden, die weltweit übernommen werden und sich zusammen mit den fortschreitenden KI-Fähigkeiten weiterentwickeln können. Gleichzeitig müssen wir wachsam bleiben, um zu vermeiden, dass neue Formen der geopolitischen Ungleichheit entstehen, bei denen sich der Zugang zu grundlegenden wissenschaftlichen Instrumenten in den Händen einiger weniger Akteure konzentriert. Das Gleichgewicht zwischen Innovation und Sicherheit erfordert sowohl technische „Leitplanken“ als auch eine integrative Steuerung. Nichtsdestotrotz ist es jetzt an der Zeit, diese Rahmenwerke zu schaffen.
Durch ihre geplante „Biotech and Biomanufacturing Initiative“, die manchmal auch als „EU Biotech Act“ bezeichnet wird, unternimmt auch die EU Schritte, um die Steuerung der Biotechnologie zu formen. Im Juni 2025 hatte die Europäische Kommission eine strategische Mitteilung veröffentlicht und Konsultationen eingeleitet, aber noch keinen formellen Gesetzesvorschlag veröffentlicht. Die Initiative konzentriert sich zwar in erster Linie auf Wettbewerbsfähigkeit, Innovation und die Verringerung des regulatorischen Aufwands für Biotech-Startups, sie enthält aber auch Verweise auf Sicherheit, Standardisierung und die Widerstandsfähigkeit der Lieferkette. Insbesondere haben einige Interessengruppen die Annahme eines EU-weiten Know-Your-Order (KYO)-Rahmens für sensible biologische Materialien und Ausrüstung gefordert. Expert*innen der Gemeinschaft für Europäische Forschung und Innovation im Dienste der Sicherheit (Community for European Research and Innovation for Security) haben Bioterrorismus als eine der zehn wichtigsten Sicherheitsprioritäten identifiziert. Trotz eines breiten Bewusstseins für solche Risiken gibt es derzeit keine EU-weit einheitliche Vorschrift für die Überprüfung von Bestellungen von genetischem Material oder von risikoreichen Laborinstrumenten. Die Policy-Organisation Pour Demain hat die Bedeutung solcher Maßnahmen hervorgehoben und argumentiert, dass ein EU-weiter KYO-Rahmen unerlässlich ist, um den Missbrauch von biologischen und KI-gestützten Designfähigkeiten zu verhindern. Es bleibt abzuwarten, ob diese Elemente in künftige Regelwerke einfließen werden, aber die Initiative könnte eine regulatorische Grundlage für die Überwachung von KI-gestützten Biodesigninstrumenten bieten.
- Reis, K. (2024). Technologische Implikationen von KI für biologische Risiken. Göttsche, M., & Daase, C. (Hrsg.), Perspektiven auf Dual Use. CNTR Monitor – Technologie und Rüstungskontrolle 2024. PRIF – Leibniz-Institut für Friedens- und Konfliktforschung. https://monitor.cntrarmscontrol.org/2024/technologische-implikationen-von-ki-fur-biologische-risiken/ ↩
- Jumper, J., Evans, R., Pritzel, A. et al. (2021) Highly accurate protein structure prediction with AlphaFold. Nature, 596, 583–589. https://doi.org/10.1038/s41586-021-03819-2 ↩
- Carter, S. R., Wheeler, N. E., Isaac, C. R., & Yassif, J. (2024). Developing Guardrails for AI Biodesign Tools. Nuclear Threat Initiative. https://www.nti.org/analysis/articles/developing-guardrails-for-ai-biodesign-tools/ ↩
- Paterson, A. (2024, 14. November). [Vortrag bei Webinar]. In Nuclear Threat Initiative, Developing Guardrails for AI Biodesign Tools. https://www.nti.org/events/developing-guardrails-for-ai-biodesign-tools/ ↩
- Chen, Y., Hu, Z., Wu, Y., Chen, R., Jin, Y., Chen, W., & Huang, H. (2024). Enhancing Biosecurity with Watermarked Protein Design. bioRxiv. https://doi.org/10.1101/2024.05.02.59192 ↩